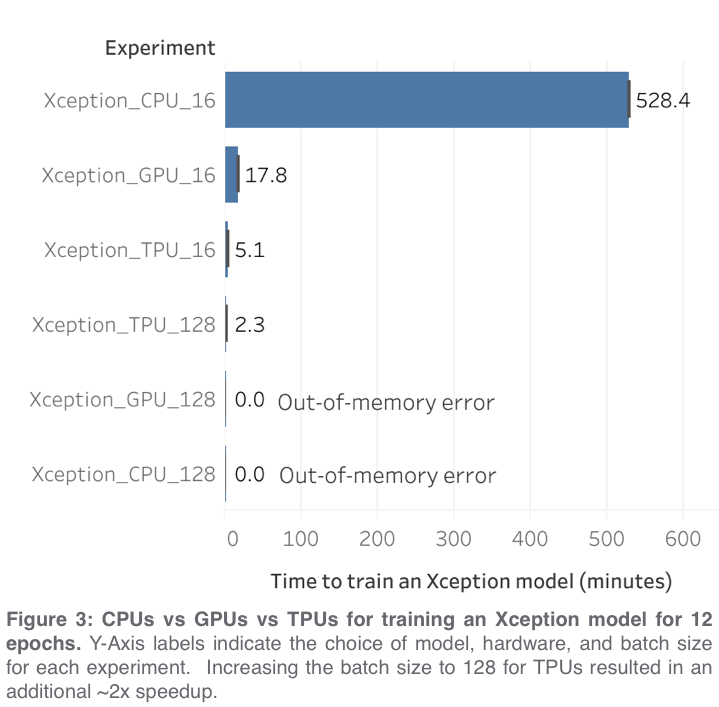
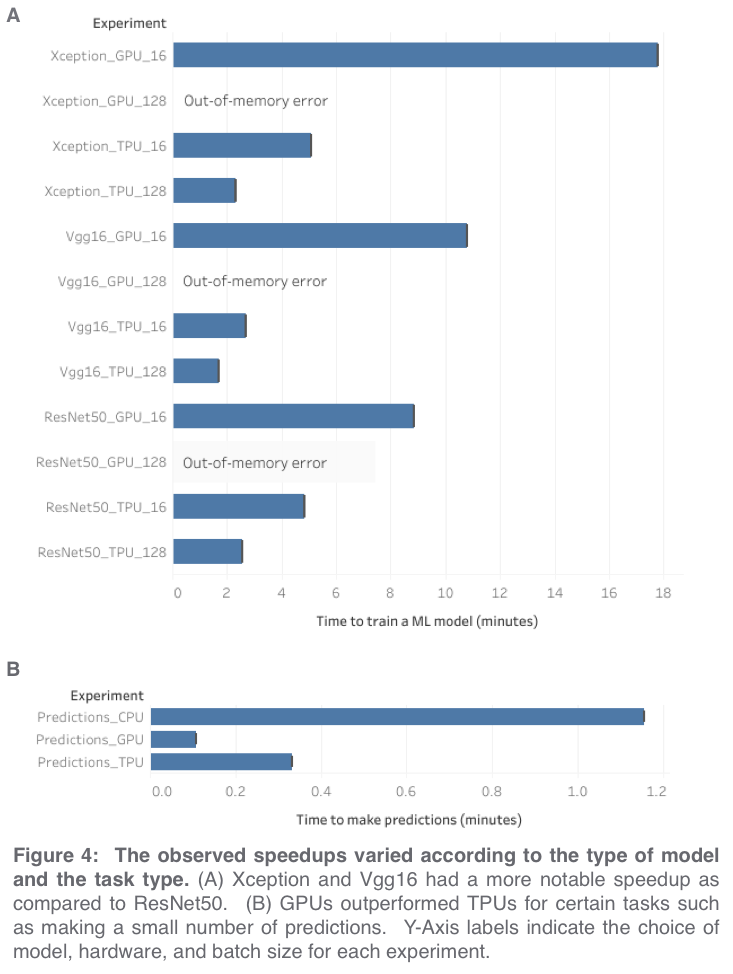
Kggle散歩(October 2020)
10月は、OpenVaccine(10/5:訂正10/6)とOSIC(10/6)とRSNA(10/26)が締め切りを迎える。
これらのコンペの中からメダルを1個獲得できれば、2個目となり、Expertにランクアップされる。
しかし、どのコンペも難しい。
OpenVaccine:メダル圏の外ではあるが近くにいる。(10/2追記:すごい勢いで遠ざかっていく、public LBでは勝負にならない)
OSIC:適正なパラメータセッティングの方向性が見えない。
RSNA:いまだに、戦う手段を持っていない。(OpenVaccineとOSICが終わってから検討する)
10月1日(木)
OpenVaccine:
アンサンブルの重みの検討:
最大値と最小値の差が2/1000程度ある6件のデータを用いたアンサンブルでは、単純平均の場合と比べると、重みづけにより、public LBは、2/10000程度改善した。
今の自分の順位だと、周辺のチームは数時間で2/10000くらい上がってしまうので、もっと効果的なスコアアップ手段がないとメダル圏内は難しい。
少しスコアが悪くても、スコアが近いものを複数合わせると良くなるので、今日検討した6件以外のデータについて、アンサンブルに使えるかどうか、明日、調べる。
10時間くらいで、メダル圏内との距離が、5/10000以上、大きくなった気がする。
13時間くらい前に、新たなコードが公開された。活発な議論が展開されている。
伝説のKagglerまで現れてコメントしている。(ただし、ネガティブなコメント?)
それにしても、参加していないようでも、ちゃんと、見ているんだね。
しかし、非常に難しい状況になってきた。
10月2日(金)
OpenVaccine:
メダル圏との差が2/1000以上になった。
チャレンジしないと、とても追いつかないし、追いつけない戦いはしたくないし、ここであきらめることもしたくない。(public_test_dataが9%と少ないので、どんでん返しにも少しは期待しながら・・・。)
まずは、アンサンブルの効果確認から始めよう。
メダル圏内が、0.239+となっているときに、3件の0.247+と4件の0.246+を利用できるかどうかを調べるのは無意味のように思うが、やってみよう。
それぞれの効果はそれなりに認められたが、最終結果への効果は、4/100000であった。
順位変動なし、というより、この作業中にも順位は下がった。
自分のアンサンブルのやり方では、public LB値は、最もよいモデルのそれよりも、2/1000くらい小さくなることがわかった。
自分の単一モデルの結果0.243+は、現在トップの0.228とは、6-7%くらい違う。
efficientnetの'b0'と'b7'くらい違うということか。
mrkmakr氏の公開コードの概要説明を転記させてもらおう。
training scheme
(1) train denoising auto encoder model using all data including train and test data
(2) from the weights of denoising auto encoder model, finetune to predict targets such as reactivity
rough network architecture
inputs -> conv1ds -> aggregation of neighborhoods -> multi head attention -> aggregation of neighborhoods -> multi head attention -> conv1d -> predict
this architecture was inspired by https://www.kaggle.com/cpmpml/graph-transfomer
最後にinspireされたプログラムとしてあげられているのは、CPMP氏の公開コードで、NFL Big Data Bowlコンペのものである。
(さきほど、スカパーのチャンネルを適当に変えていたら戦争映画にでくわした。途中からだったが、引き込まれて、最後まで見た。Darkest Hourというタイトルだった。)
10月3日(土)GPU:0/38, TPU:0/30
OpenVaccine:245/1543, 0.24148, 0.23846
現時点で、3/1000の改善が必要。
単独のスコア0.24353を0.24053まで下げる方法を探す。
最初に試した結果も、2番目に試した結果も、散々(0.245+)だった。
深夜までトライしていたが、0.24353すら超えられない!
OSIC:309/2063, -6.8072, -6.8070
検討中
10月4日(日)
OpenVaccine:
新たに得た3件の予測データをアンサンブルに追加したが、スコアは改善されなかった。
ともかく、最良と思われる結果を、締め切りまで、計算し、submitしつづけるだけだ。(やけくそ!)
ようやく単独で0.242+となったが、ちょっと遅かったかな。
メダル圏内はすでに0.237+にまで下がっているので、このスコアでは、届かない。
計算資源を節約しながら(時間制限を受け入れながら)ハイスコアを出すには、まだまだ、技術が足りないということだ。
10月5日(月)
OpenVaccine:
昨夜計算した結果はcommit中のトラブルにより失ったため、計算は終了した。あとは、新たに得た2件の結果をアンサンブルに追加してsubmitするだけである。
今、public LBで320番くらいで、新しい結果をどう組み合わせても、メダル圏内に入ることはないが、最後の5回のsubmissionで最善をつくそう。
コンペ挑戦終了。
0.24105, 311st(10/5/10:57)
OSIC:
締切日の前日にして、GPU割当時間を使い切った。
といっても、結局、方向が定まらず、気になる計算条件を煮詰めようとして計算を繰り返していたが、見えない相手に対して、どう攻めたらよいのか、わからないままである。
88回のsubmit履歴が残っているが、どのモデルがprivate test dataに対して最も確からしい予測結果を出せるのか、判断基準がわからないままである。
コンテストのために用意されたデータではなく、現実世界で使われているデータで、よく整理されたデータというよりは、医療現場で蓄積されたデータを取捨選択することなくかき集めてきたもののように思う。
それだけに、このデータから何が言えるのかを根拠をもって説明できるだけのモデルでなければならないのだろうが、残念ながら、そこには辿りつけていない。
10月6日(火)
OpenVaccine:
最終提出期日を間違っていた。
昨日ではなく、今日だった。
OSIC:
今日が投稿最終日だ。
RSNA:
20日間は、このコンペに集中しよう。
現在、330チームがリーダーボードに掲載されている。
A pulmonary embolism (PE) is caused by an artery blockage in the lung.
Currently, CT pulmonary angiography (CTPA), is the most common type of medical imaging to evaluate patients with suspected PE.
In this competition, you’ll detect and classify PE cases.
10月7日(水)
OSIC:
暫定結果は、1504番目
気合十分で始めたコンペだったので、この結果は、非常に残念である。
結局、最後の2回のsubmissionを選んだ。
public LBの最も良い値を選んでいたら、1120番くらい。
submitした中で最も良かったのは、最終順位で106番で、銀メダル相当だった。これは、NNのフィッティング結果が良かったのでsubmitしたのだが、public LBは良くなかったし、これを選ぶ特段の理由がなかったので、選ばなかった。
これは、順位が大きく変動するだろうという予想どおりとなったが、どうすれば良いか、どれを選べばよいかは、わからなかった。
OpenVaccine:
暫定結果は、379番だった。
メダル圏外ということでは、残念な結果だが、これは、ほぼ、予想通りである。
普通に計算して、その結果をアンサンブルしただけなので、大きく上がることも下がることもないだろうと思っていた。
RSNA:
データが非常に大きく、inferenceに長時間を要することから、コードコンペでありながら、inferenceに限るということだが、こういうのは、自分にとっては初めてのことである。
画像の扱いも、少し、特殊であり、train画像の枚数も多い。
データは、大きすぎて(900 GB以上)ノートパソコンにダウンロードできない。
10月8日(木)
RSNA:379チーム
今日は。trainコードとinferenceコードを準備しよう。
参加者が少ないためか、公開コードが少ない。
trainコードの準備
・学習させたモデルはsaveする。
・saveしたモデルは、kaggle datasetとして保存する。
inferenceコードの準備
・ 学習済みモデルのload:作成したkaggle datasetから学習済みモデルを読み込む。
これだけのことなんだが、モデルの保存・読み込みは、これまで、それほど意識して使ったこなかったが、これを機に、使えるようにしよう。
モデルの保存等に関するコード:A. Geronさんのテキスト314ページ
Saving and Restoring a Model
When using the Sequential API or the Functional API, saving a trained Keras model is as simple as it gets:
model = keras.models.Sequential([...]) # or keras.Model([...])
model.compile([...])
model.fit([...])
model.save('my_keras_model.h5')
Keras will use the HDF5 format to save both the model's architecture (including every layer's hyperparameters) and the values of all the model parameters for every layer (e.g. connection weights and biases).
It also saves the optimizer (including its hyperparameters and any state it may have).
<雑談>
AIを極めることに取り組んできたが、そこから遠ざかっている自分の姿が見える。自分に見えているAIは、自分が見てきた過去のプログラミングでは成し得なかったことを成し遂げているが、それらが、ものすごく大きなものに見えることもあれば、想像していたものとは全く違って見えることもある。 (twitterに書こうとしてやめた)
今のプランは、メダル獲得競争と、メダル獲得を強いモチベーションにして、様々な課題解決のためのプログラミング技術を習得しながら技術レベルを高め、様々な課題に対してより高いレベルの解決方法を模索していくことであり、その先に、次の世代のAIを考え、構築していこうというものである。
今の自分の最大の弱みは、英語力である。このハンディーは非常に大きい。マニュアルをいちいち和訳していては進まないし、プログラムは英語そのものだから、プログラムもマニュアルも論文も書籍も斜め読みできる英語力がなければだめだ。
そう思って、昨年の6月頃にdeep learningを勉強するためのテキストとして、F. Chollet氏の著書を購入し、その後も、何冊か英語のテキストを購入してきた。しかし、読まなければ、書かなければ、使わなければ、英語力は身に付かない。
気がつくと、Google翻訳やオンライン翻訳に頼っている自分がいた。英語のテキストが理解できずに和訳を買って見比べながら読むこともやってみたが、新しい概念は和訳したってわからないのだ。定義や応用事例を見てその意味を理解していくのだから、1つの概念を理解するために日本語と英語の両方でやるなんてことは非効率の極みである。
10月9日(金)
Riiid!:
Answer Correctness Prediction
Track knowledge states of 1M+ students in the wild
The goal is to accurately predict how students will perform on future interactions.
If successful, it’s possible that any student with an Internet connection can enjoy the benefits of a personalized learning experience, regardless of where they live.
面白そうだな。オンライン学習が進めば、学習プロセスに関するデータが蓄積され、各人に合った学習プロセスを提供できるようになるということか。
学習の経過がフィードバックされるようにプログラムしておけば、自律的に成長していくということか。個々人の成長に合った学習プログラムが提供できればいいな。
各人が、自分で、自分の脳にプログラミングしているために、その能力の違いが学力の違いを生じさせているのだろう。そうすると、自分で自分の脳にプログラミングできない人にとっては、このような学習システムは重要なものになるのだろう。
学力に応じたプログラムが自動的に生成されるようにすればいいのだろう。
Tailoring education to a student's ability level is one of the many valuable things an AI tutor can do.
Your challenge in this competition is a version of that overall task; you will predict whether students are able to answer their next questions correctly.
You'll be provided with the same sorts of information a complete education app would have: that student's historic performance, the performance of other students on the same question, metadata about the question itself, and more.
公開コードのパラメータを少し変えて投稿してみた。
NNよりもLGBMの方が良さそうだ。
10月10日(土)
RSNA:
10月8日に検討した結果を元に進めるが、全体の流れをチェックしやすいように、再スタートする。
1.kerasで書かれた公開コードを使ってみる。(trainを含んでいるので切り離すが、trainを含んだままでcommit->submit->scoreまで進むかどうか調べてみる)
2.コードコンペなので、internet offにしなければならないが、このコードは、'imagenet' pretrained modelのweightsを、インターネット経由でダウンロードするようになっている。
3.kaggle kernelで使うdatasetを準備する。
tensorflow/kerasのサイトの情報から、'imagenet' pretrained modelのweightsをダウンロードし、kaggle datasetに格納する。
4.このデータセットを、kaggle kernelの入力データに追加する。
5.train+inferenceプログラムを実行してcommitする。trainもinferenceも実行時間を設定しているので、各1時間で試してみる。実験なので、GPUは使わないでやってみる。
6.submitまでは無事に済んだ。
ここで、スコアが表示されるかどうかによって、次の対応が決まる。
7.スコアが出た。0.975(0に近い方が良いスコア)であった。
ということは、trainとinferenceが共存しても大丈夫ということだ。
8.GPUをonにすると、trainは5倍以上速くなるが、inferenceは2倍程度しか早くならないことがわかった。
9.試しに、512x512のデータをefficientnetB5でinferenceすると、GPUを使っても1時間に50kくらいしか処理できなかった。したがって、trainとinferenceの両方を含むコードでは、9時間の制限時間でも不足する。それだけでなく、大きなモデルで大きな画像を処理すると、inferenceだけのコードでも、制限時間内にinferenceが終わらない可能性が出てくる。ということで、trainとinferenceは分離しよう。
10.と言いつつ、0.975(これはとんでもなく悪いスコアで、トップは0.149、0.325とか0.434がたくさん並んでいる)を上回るスコアになるかどうか確かめるための計算を行う。CPUのみで、trainとinferenceのそれぞれに、4時間の時間制限をつける。0.975のときは、1時間の時間制限であった。どうなるか楽しみだが、計算とcommitを合わせて16時間、それにスコア計算の時間が加わるので、20時間以上かかりそうだ。
・こういう長い時間を要する場合は、いろんな不具合が生じて、何日もかかることがあるので、気長に、楽しみながらやるのがよい。
10月11日(日)
RSNA:
悪い予想が当たった。朝起きると、次のメッセージを残して、計算が止まっていた。
Your notebook tried to allocate more memory than is available. It has restarted.
停止していたのは、inferenceの途中だったので、train中の情報がメモリーに蓄積されていたのが、まずかったのか。ともかく、途中経過がわからないと対策のしようがない。
'view session metrics'を'on'にしておけば、メモリーの使用状態が、リアルタイムでわかる。(眠っていたらわからないけど)
途中経過を見ているところだが、Diskではない。RAMの使用量(max 16GB)が、14GBから徐々に増え、50分くらいで16GBに到達し、それ以降は、なんらかの調整機能が働いているようで、15.4-15.9GBあたりで推移している。
GPUをonにしたらどうなるのだろうか。:RAMは16GBから13GBに割り当てが減少するので直接比較できない。GPUのRAMとの関係もわからない。GPU併用との大きな違いは、CPUの稼働率が300%を超えていたのが、120%以下に下がったこと。ともかく、途中で停止せず、予定の計算を最後までやってくれたらそれでいい。
順調に進んでいるように見えていたが、one batch timeが、inferenceの進行とともに長くなっている。バッチあたり13秒くらいで始まったのが、1時間を過ぎるころには2倍くらいかかっている。
予定の3時間まであと25分のところで、同じメッセージYour notebook tried to allocate more memory than is available. It has restarted.があらわれて停止した。
嫌なパターンだ。
どうしたものか。
メモリー使用量が問題なら、batch_sizeを下げれば良かろうと思って500から250に下げると、処理時間が倍ほどかかりそうで、途中でやめた。batch_sizeが原因かどうかもわからず下げるより、上限を探ろうと1000に設定してみた。理由はわからないが、とりあえず最後まで計算が進んだので、ひとまずOK。
さらに条件検討してみよう。メモリーコントロールを学ばないといけないようだ。プログラムコード中に、del modelとか、del x,とか、見かけたような気がする。
とりあえず、inferenceもできるようになった。しかし、スコアがおかしい。最初のスコアは、一部の結果しか得られていないから仕方ないのだが、inferenceは完了したにもかかわらず、スコアがそれほど良くならない。
10月12日(月)
RSNA:
predictionの割合とLBスコア:53k/147k:0.975、88k/147k:0.934、147k/147k:0.862
train:efficientnetB0、256x256、25k-125k/1790k
いまは、predictionが進めば、スコアが少しづつだが、良くなっている(値が小さくなっている)、というレベル。
次は、predictionの後処理を調べてみよう。
evaluationを見ているのだが、よくわからん。
10月13日(火)
RSNA:
うまく動き出したと思ったのだが、submitした後のスコアリング中にNotebook Timeoutが発生した。
なぜエラーになるのかわからない。
Kaggle cources:
Kaggleから、Intro to Deep Learningの案内が来た。
昨年、deep learningを本格的に勉強し始めたころは、Kaggle minicource ?、には、たいへんお世話になっていたのだが、もの足りなくなって、data campを利用したり、テキストを追加購入したりして勉強していたのだが、ある程度習得したつもりで、あとは、応用だとばかりに、Kaggleコンペにのめりこんでいるところである。
しかしながら、課題に取り組んでみると、自作のモデルでは、良い性能が出せなくて、いつしか、公開コードをフォークするのがあたりまえのようになってきた。
こういう状況をみすかされたわけではないと思うが、良い機会ととらえ、勉強してみようと思っている。
昨晩は、YouTubeでDeep Learning State of the Art (2020) | MIT Deep Learning Seriesを聴講したが、お話は、いくら聞いても身に付かないと感じていたところである。
入門レベルだが、英語の勉強を兼ねて、取り組もう。
10月14日(水)
mail from Kaggle:
You’re not too far from receiving a completion certificate!

For every course you complete on Kaggle, we’ll issue an official certificate that celebrates the progress that you’ve made in your data science and machine learning journey.
手を付けたはいいが、ほったらかしにしていたら、こんなメールが届いた。
今月中に、全コースの終了証をもらえるように、がんばってみよう。
<雑文>
ここ2,3日、頭が重い。なぜかと考えてみた。脳になんらかの負担がかかっているということなんだろうな。脳には、体のあらゆる情報が集まっているのだから、頭が重いのは、体のどこかに不具合が生じているということだろう。ここ2,3日の変化と言えば、1週間くらい前から、ダンベルを持ち出して、腕力のトレーニングを始め、数日前からは、push ups(腕立て伏せ)の回数を増やそうとして、計算の合間に、強めのトレーニングをしてきた。今日は、肩から上腕部にかけて痛みがあり、同時に、頭の重さを強く感じるようになった。こうなると、トレーニングをやめてしまい、三日坊主に終わってしまうことになるんだろうな。負荷を調整しながら、トレーニングを続けよう。
RSNA:
まだ、trainとinferenceの両方を含むプログラムを使っている。
性能アップのためには、十分なtrainingが必要であり、trainとinferenceを分離する必要がある。これが、まだうまくいっていない。
train modelは、detasetに入れて、inference notebookの入力データに追加して、読み込もうとしているのだが、・・・。
今日は、trainとinference共存で、trainをかなり多くしてみたところ、inferenceに移ってからの処理速度が遅くなった。負荷がかかってるなという気がしてから30分くらいの間にプログラムが停止した。
GPU使用、'B0', 256x256で3エポックでも18時間くらいかかりそうなので、もうこれは、単にtrainとinferenceを切り離せばよいというものではないことを確信した。
ここからメダル圏内に入るために、trainにTPUを使おうと思う。当初からそうしないとだめだろうなというところにきてしまった。
ぼちぼち勉強しながら前進しよう。
Winner's Interview:Tweet Sentiment Extraction
Kaggleからのメールを見て、インタビューの一部を聴講してみた。2位は日本人3名のチームだった。説明内容もトークも良かった。あたりまえだが、レベルが高い。雲の上の人たちだなと感じてしまう。
intro to machine learning:
ぼちぼち勉強中。
10月15日(木)
RSNA:
スコアの良いコードが公開されていたので、利用させていただいた。それでもメダル圏外である。公開したチームは、(当然)それより良いスコアで上位にいる。
関連するdiscussionから、trainには、相応の計算資源と計算時間(+コーディング+デバッグ)が必要であることがわかる。
trainに用いたコード(例)も公開されている。
この公開コードによって、Project MONAIの存在を知った。
https://docs.monai.io/en/latest/:以下、ここから引用
Project MONAI
Medical Open Network for AI
MONAI is a PyTorch-based, open-source framework for deep learning in healthcare imaging, part of PyTorch Ecosystem.
Its ambitions are:
-
developing a community of academic, industrial and clinical researchers collaborating on a common foundation;
-
creating state-of-the-art, end-to-end training workflows for healthcare imaging;
-
providing researchers with the optimized and standardized way to create and evaluate deep learning models.
Features
The codebase is currently under active development
-
flexible pre-processing for multi-dimensional medical imaging data;
-
compositional & portable APIs for ease of integration in existing workflows;
-
domain-specific implementations for networks, losses, evaluation metrics and more;
-
customizable design for varying user expertise;
-
multi-GPU data parallelism support.
Modules in v0.3.0
MONAI aims at supporting deep learning in medical image analysis at multiple granularities. This figure shows a typical example of the end-to-end workflow in medical deep learning area: 
MONAI architecture
The design principle of MONAI is to provide flexible and light APIs for users with varying expertise.
-
All the core components are independent modules, which can be easily integrated into any existing PyTorch programs.
-
Users can leverage the workflows in MONAI to quickly set up a robust training or evaluation program for research experiments.
-
Rich examples and demos are provided to demonstrate the key features.
-
Researchers contribute implementations based on the state-of-the-art for the latest research challenges, including COVID-19 image analysis, Model Parallel, etc.
The overall architecture and modules are shown in the following figure: 
これまでに、MONAIを見たり聞いたりしたことがない。情報収集力に問題ありだな。
0.1.0-2020-04-17となっているので、半年前に公開されたということか。
PANDAとTReNDSのdiscussionでは話題になっていたようである。
特に、TReNDSでは、discussionで7件、notebookで1件、検索でひっかかった。
discussionで多くでてきたのは、上位者が入賞記事でMONAIに言及していたため、すなわち、上位入賞者はMONAIのことを4か月以上くらい前から知っていて使っていたのである。さすがだな。
Kaggleは、100 ppmレベルの違いを競っていて馬鹿らしく見えるときもあるが、さらに上を目指すために、新しい優れた技術を探し当てて利用し、あるいは、それらを創り出すきっかけになっているのだろう。
Intro to Machine Learning:
初歩の初歩だけど、あらためて、やり直してみると、最近はプログラムを一から書いていないからだろうな、解説やコード例を何度も見返さないと正しいコードが書けなくなっている。
10月16日(金)
Intro to Machine Learning:
本日22時46分、やっと終了した。過去にsubmitまでやった履歴が残っていたが、記憶に残っていたのは少なかった。
Bonus Lessons:Intro to autoML:
google cloud autoMLの宣伝?ひとまず中断!
Intermediate Machine Learning:
2019年9月14日に終了していた。
記録が残っているのだが、記憶に残っていない。
気がつけば、ボケ老人になっていたということか。
1年前は、3時間で片付けようとか、3日でやってしまおうとか、1週間もあればできるとか、3か月後にエキスパート、6か月後にマスター、1年後にグランドマスターとか、言っていた。
そのようなノリで、このコース:Intermediate Machine Learning:も、斜め読み(英語の斜め読みはいい加減だったと思う)とコピペで終了したのかもしれない。正確には思い出せないが、じっくりと腰を据えて取り組んだものではなさそうだ。
PythonがYour Completedの中に入っていたので、中を見たが、とても理解できているとは思えない。とにかく、Coursesを、全部、やりなおそう。
10月17日(土)
RSNA:
パラメータを変えてRun, commit, submit, スコアに変化なし。
Cources:Pandas
英文の単語の配置を変更して少し記号を追加すればコードになるのに、日本語が間に入ってしまうから、難しくなる。それだけのこと。
Lessonは、TutorialとExerciseに分かれていて、Tutorialのテキストを読み、Exerciseの各問題を解く(コードを入力する)、ということだと思っていて、Tutorialは読むだけだったが、Editorを動かせば、例題のコードを好きなように変更して、コードの動作確認をしながらコーディングを学ぶことができる。いまごろ気付いた。
10月18日(日)
RSNA:
進捗なし。
Cources:Pandas
Create variable centered_price containing a version of the price column with the mean price subtracted.
(Note: this 'centering' transformation is a common preprocessing step before applying various machine learning algorithms.)
centered_price = reviews.price - reviews.price.mean()
Exercise中にYour notebook tried to allocate more memory than is available. It has restarted. このメッセージが現れた。全く、理由がわからない。
リスタートしたら、こんどは、このメッセージがすぐに現れて、停止した。
Lyft:
フォークして実行するだけ。だめだな。進歩無し。
10月19日(月)
今日は、朝から、Cheatingに関する議論を眺めている。
Cheatingは不正行為全般を指しているので案件ごとに内容が異なり、対応も異なるが、詳細を記することは、それを真似た不正が生じる恐れもあり、対応策にしても明確に示すのは難しく、検討のさなかにあるということである。
Plagiarismの話題では、盗作といえば、公開コードに関するものだが、公開コードを使って計算して投稿すること自体が問題だという人もいる。これを許容していることがKaggleの特徴であり、これ自体は問題ではない。
問題は、Kaggleのランク付けと関係しており、公開コードのupvote数およびその件数によってランクが上がり、コンペのランクと同じ呼称、エキスパート、マスター、グランドマスターが与えられる。この公開コードでランクアップを狙う過程で盗作があるということである。コードを販売して利益を得ているのではないが、公開コードへの投票が自分への投票になるということにおいて、栄誉という利益を得ていることになり、それが、著作権を盗んでいることになるのである。
論文を書いたことのある人はわかると思うのだが、著作物は、そこで使われている文章、図面、手段、理論などの出典を明らかにすることと、著作者の許諾を得ることは必須であるが、それを理解しない人が多いということである。
こういう決まり事は、体得していないとわからないものである。SNSで発信する場合も同じ縛りがあるのだが、個人使用と公開との境界があいまいになり、放置されていることが多い。
Plagiarismに関する議論で、誰かが、盗作者に対しては、きちんと取り締まるべきであり、盗作か否かの境界を明確にし、担当者を増やすなりしてきちんと対処してほしいということをKaggleに対して要望していた。これに対して、Kaggleの担当者の1人が、そのとおりであるが、全体のバランスも勘案しながら良い方法を検討していくと述べた。実社会でもそうだが、訴えがなければ、裁判は始まらず、訴えができるのは、原則として、損害を被った当事者であることから、利害関係のない人から盗作の指摘があっても、主催者が事実関係を調べるべきである、ということにはならない。
コンペは賞金が絡むので不正を見逃すことはできないが、それ以外は、当事者がKaggle参加者であれば、ゆるくしておいてもよさそうな気はする。
最近、GitHubから引用した人が引用元を明示していなかったことを、制作者に指摘されているのを見た。これは非常識すぎると思った。GitHubには、ライセンスの種類が明記されており、表示が必要な場合には、表示の仕方が明記されている。
RSNA:
STARTER DATASET: Train JPEGs (256x256)
This competition is challenging due to the huge dataset size. In addition, people may not be familiar with medical imaging and CT scans.
To lower the barrier to entry to this competition and to encourage more people to participate, I have converted all of the DICOM images into JPEGs (about 52GB), 256x256 pixels (standard is 512x512).
GMのIan Pan氏が、1か月も前に、コンペに参加してもデータが大きすぎて、手も足も出ない者に対して、このようなデータセットを作っておられたことに気付かなかった。
Cources:Pandas
学習中のエラーの原因は、コードの書き間違いだった。'.'とすべきところに'/'が入っていたのが原因だったようだ。しかし、メッセージの意味とのつながりがわからない。
とりあえず、エラーは解消し、次に進めるようになった。
今日のスロージョギング:4.2 km, 46 min
10月20日(火)
Cources:Pandas
ようやく、一通り終えた。TutorialとExerciseがペアになっているので、Tutorialで学び、Exerciseで学んだことを確認するものと思ったが、そうではない。両方を合わせて1つであり、両方合わせても、Pandasのごく一部にすぎない。ということは、このレベルのことは、スイスイできるようになっていなければならないということだろう。
Cources:Data Visualization
Progressが53%になっているので半分くらいやったのかなと思ったら、Tutorialをすべて眺めただけだった。ということで、Exerciseにとりかかろう!ではなくて、Tutorialの最初から、英文で内容を理解しよう!
genre:ゲンレと発音していて、ジャンルの意味だとは知らなかった。こんなレベル!
categorical scatter plot:
sns.swarmplot(x=candy_data['chocolate'], y=candy_data['winpercent'])
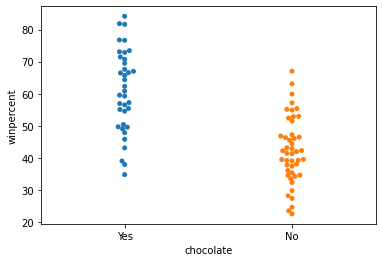
作図は形式的にやっていても得るものは少ない。表から直観的に概要を捉えたうえで、仮説を立てながら、図示し、図から関係性を定量的につかんでいく、というような感じで進めていくことができなければ、コードも頭に入らない。1年くらい前に取り組んだ時には、表の意味を理解することに、ものすごく手間がかかって、結局、放り出していたのだろうと思う。
RSNA:
何かやれることはないかと考えていただけである。
10月21日(水)
自主学習:ニューラルネットワークと第一原理計算
最近の状況をA4半ページ以内のスペースで紹介することが直接の目的。
引用論文は次の3件にするつもり。
1) D. Pfau, J. S. Spencer and A. G. D. G. Matthews, Phys. Rev. Res. 2, 033429 (2020)
Ab initio solution of the many-electron Schrödinger equation with deep neural networks.
2) J. Hermann, Z. Schätzle and F. Noé, arXiv:1909.08423v5 [physics.comp-ph] 23 Sep 2020
Deep neural network solution of the electronic Schrödinger equation.
3) K. Choo, A. Mezzacapo and G. Carleo, Nat. Commun. 11, 2368 (2020)
Fermionic neural-network states for ab-initio electronic structure.
1番目の論文では、FermiNet、2番目の論文ではPauliNet、という略語が用いられているが、3番目の論文では、どちらの略語も使われていない。
RSNA:
2~3日、見ていない間に、ハイスコアのコードが公開されたようだ。
とりあえず、フォークさせていただいて、実行してみる。
commit, submitでスコアが出ると、そのスコアを上げる方法はないものかと思い、コードをながめまわすことになるのだが、このときに、経験値が生きてくる。
ハイレベルな人たちは、自分のコードと比較して良いところを取り込むだろうし、組み合わせの上手な人は、いろいろな公開コードから使えそうなパーツを借りてきて、組み上げていくんだろうな。このときに忘れてはならないのは、それぞれのパーツのオリジナルの引用元を明示しておくことだな。オリジナルが分からない場合でも、最低限、直前の引用元を明示しておくことが必要だな。
Kaggleのmicro courceを少し勉強しただけでも、コードは理解しやすくなるものだし、フォークしたコードのチューニングを繰り返している間も、少しづつはコードの理解が進んでいくものだ。
時にはレベルは低くても自前のモデルにこだわるのもいいし、時には、ベースモデルに、パーツを組み合わせていくのもいいし、ハイレベルのモデルからコーディングを学ぶのもいいし、いろんな使い方をして、前進していけばいいんだ。
とにかく、立ち止まらないことが大切だろうな。
10月22日(木)
RSNA:
昨日計算した結果は、timeout errorであった。パラメータの値の変更が裏目に出たようだ。公開コードの理解を進め、改善の可能性を探る。
自主学習:ニューラルネットワークと第一原理計算
4) Z. Schätzle, J. Hermann and F. Noé, arXiv:2010.05316v1 [physics.comp-ph] 11 Oct 2020
Convergence to the fixed-node limit in deep variational Monte Carlo
・PauliNetとFermiNetの違い(同一内容の異称かどうか調べる)
・variational quantum Monte Carloからの改善点と発展性
45年くらい前に最も興味のあった分野なんだけど、言葉に慣れても、量子化学計算の式の理解力が不足しているために、途中で思考が途切れる、あるいは、停止する。それでも、当該分野の最近の展開を、インパクトのある文章に仕上げなければならないのだ。
10月23日(金)
RSNA:
メダル圏内が見えてこない。
micro-cource:Data Visualization
micro-cource:Data Cleaning
Exerciseのデータベースが、building permits(建築許可)である。こんなデータに興味はない。そうすると内容を考えずに形式的に解答してしまう。やる気が失せることもある。NaNとなっている理由が理解できなければ、その次の処理の方法とその理由を理解することが難しくなる。データの意味が分からないと、そのカラムを無視して良いのか、仮のデータを代入すべきかなどを正しく判断できない。
Tutorialのデータはnfl_dataで、これも、面白いものではない。そのために、つい、読み飛ばしたり、内容を理解しないままに形だけ真似てしまって、必要なコードが頭に入っていなかった。Kaggleのコンペではよく出てくるようだ。アメフトのルールをある程度知らないと理解できないことが多い。
好き嫌いや興味の有無は、技術習得の段階では、邪魔者でしかない。
興味が無くても、その内容を理解して、必要な技術を1つづつ習得していかなければならない。
10月24日(土)
RSNA:
final submission deadlineまであと3日となった。5日前に公開されたコードのLBスコアは、100番から320番まで並んでいる。3日前は、およそ、60番くらいから260番くらいまで並んでいた。ということは、3日間の間に、40名くらいが、0.233を超えていったということになる。
trainからやりなおせるなら、画素数を増やして、EfficientNetB0をB5以上にするとか いうことになるのだろう。
ハイスコアの公開コード、5回ほど、パラメータを変えて計算してみたが、予測データに変化がなかった。ようやく6回目にその値が変わったので、結果を楽しみに、commitしていたら、並行してmicro-courceのexaciseをやっているときに、パソコンがフリーズした。1時間くらいして復帰したが、動作不安定となった。commitは終了していて、submitまでできたが、スコアが出るかどうか不安。
ほぼ同じ条件で、再計算中。---> エラー発生(上記エラーの影響だろうな:windows10の性能が悪いということだろうな:4時間半のロス)
同一条件で再計算。結果は、0.696と、ひどいものだった。悪化させただけだ。
先ほど、別の条件で計算している途中で、あと1時間でメンテナンスのためコンピュータを停止するからcommitするようにとのメッセージがあった。
計算途中でcommitするとはどういうことなのだろうか。
途中の状態が維持されて、その状態から再開されるというのだろうか、やったことないからわからないけど、うまくいきそうに思えない。
何か仕掛けがあるのかどうか、どこかで時間をとって、調べてみよう。
micro-cource:Data Cleaning:scaling and normalization
# for Box-Cox Transformation
from scipy import stats
# for min_max scaling
from mlxtend.preprocessing import minmax_scaling
in scaling, you're changing the range of your data
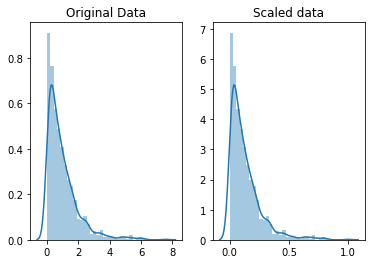
in normalization, you're changing the shape of the distribution of your data.
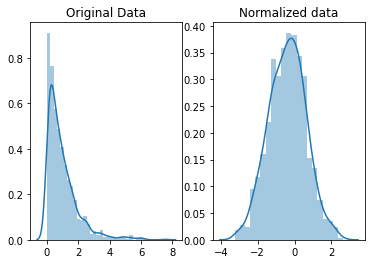
これは、非常にわかりやすい説明だ。
重要なのは、スケーリングや規格化をいつ、どこで(どのような場面で)、どのように使うかということだろうな。
micro-cource:Data Cleaning:parsing dates
earthquakes['date_parsed'] = pd.to_datetime(earthquakes['Date'], format="%m/%d/%Y")
よくわからんが、10/24/2020 とか、2020-10-24とかの期日の表示方式に関すること。
micro-cource:Data Cleaning:character encodings
# helpful character encoding module
import chardet
UTF-8 is the standard text encoding. All Python code is in UTF-8 and, ideally, all your data should be as well. It's when things aren't in UTF-8 that you run into trouble.
Try using .decode() to get the string,
then .encode() to get the bytes representation, encoded in UTF-8.
micro-cource:Data Cleaning:inconsistent data entry
import fuzzywuzzy
from fuzzywuzzy import process
これは、かなりたくさんのデータを、手作業で眺めてみて、問題点を拾い出すことを丹念にやって、見つかった課題を、自動で正確に処理するために、便利な関数を使うことができるようにするということのようだ。
10月25日(日)
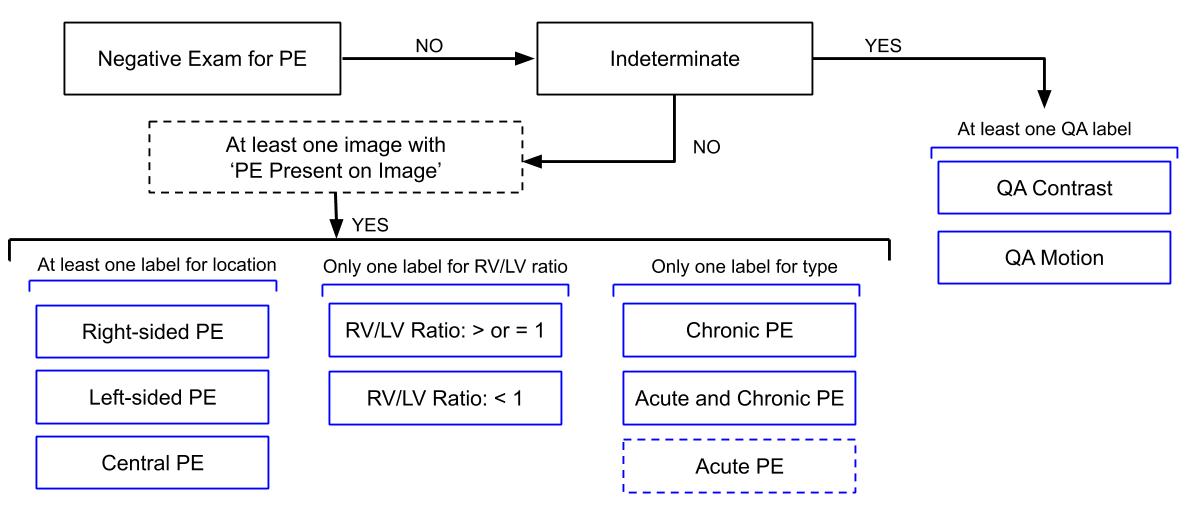
RSNA:
Data setの理解:数%
Metricの理解:数%
モデルの構築:ゼロ
目標達成度:ゼロ
たとえば、データの説明のために、わざわざ次の図が掲載されていたのだが、それぞれの項目の意味とともに理解しておかないとプログラムが作れないことが分かっていながら、理解しようとしなかった。もちろん、容易なことではない。でも、それをやらないと、この先はない。
<行事メモ>
「AIを用いた画像処理・認識技術の進展」
Zoomウエビナー
4件の講演があった。最も新鮮だったのは、AIチップの話である。そのほかの話は、わかりやすかったが、驚きはなかったので、省略する。
開発中のAIチップは、コンタクトレンズに組み込み、涙で発電してAIチップを駆動し、涙の中の糖分の量の情報を得て、それを電波発信してスマホ等で受信するというもので、類似のシステムは、Googleで開発が進められていた(採用している技術は本質的に異なるところがある)が、中止されたとのこと。
AIチップは、最先端の半導体デバイス工場でしか生産できない。すでに衰退している日本の状況が心配である。AIチップの話をされた名古屋大学の先生は、設計したチップの製造を台湾のTSMC社に委託しているとのことである。悲しい話ではないか。
クラウドはもはや時代遅れで、あるべき姿はエッジということかもしれない。それも、AIマシン専用の電源が必要な設備ではなく、パソコンレベルでもなく、スマホレベルでもなく、ゆくゆくは、チップレベルということになるのか。
AIチップの開発は、世界中で競争が激化しているようだ。先を走っていたNVIDIAやGoogleも安閑とはしていられないくらい多くの企業が参入しているようだ。
我に返ると、TPUを使うプログラムを書いたことがないというのは、もはや、時代遅れということになる。なんとかしないといけない。
<雑談>
コンペへの取り組み方:
これまでの取り組み方の全てを用い、どれかを捨てるということはしない。捨ててきたものとして、「ゼロからモデルを組み立てる」というのがある。これを捨ててきた理由は、コンペによって得るものが殆どない、すなわち、自前のプログラム作成に固執すると、プロや研究者の高度なプログラミング技術を学ぶ機会が殆どないまま、完成度もレベルも低いコードをつくることに終始していたことから脱却したいとの思いを強くしたからである。
しかし、公開コードをベースにしてコンペに参加すると、自らコードを作り上げていく際に必要となるデータの扱い方、metricsの理解とそのコードへの反映、特徴量の理解、判断、抽出方法など、課題毎に異なる様々な状況や要因を把握し理解する能力を養い向上させる機会が得られないなど、マイナス要素が大きく、プログラミング能力向上の機会が大きく損なわれている。
Kaggleコンペから、エキスパート、マスター、グランドマスターに認証されるだけの事をなす能力を獲得することは容易ではなく、重要であり、その過程で学ぶものは多いと予想されるが、その過程で獲得していくさまざまな技能や技術は、基本的には、他者が開拓、開発したものであることが大半であろうと推測される。
そのレベルに達してから考えるべきことかもしれないが、そのレベルに達するための道は複数あって、どの道をたどるかによって、同じランクであっても、質的に異なることは想像に難くない、というか、コンペに参加してきた経験から、容易に想像できる。
自分がそうありたいと思うあるべき姿は、明確に、かつ、継続的に意識し続けなければならない。それは、資格、技能、技術、理論、哲学、倫理、思想、のどれにも偏ることなく追求し続けていきたいと思う。息絶えるまで。
micro-cource:Computer Vision
In this course, you'll:
- Use modern deep-learning networks to build an image classifier with Keras
- Design your own custom convnet with reusable blocks
- Learn the fundamental ideas behind visual feature extraction
- Master the art of transfer learning to boost your models
- Utilize data augmentation to extend your dataset
If you've taken the Introduction to Deep Learning course, you'll know everything you need to be successful.
1. The Convolutional Classifier
2. Convolution and ReLU
ReLUは指示されたとおりに使っていただけだったが、ここでの説明と、処理例をみて、納得した。フィルター処理したときには、特徴は浮かび上がるものの、境界がぼやけて見えるのだが、ReLUを通すと、境界が明瞭になる。それは、フィルター処理の後にReLU処理すると行列の負の値がゼロになるためだということで、それが境界を明瞭にしているということがよくわかった。
3. Maximum Pooling
2x2のmaximum poolingを学んだときに、average poolingも紹介されていた。maximum poolingだと、位置がずれる。直観的に、average poolingの方が良いのではないかと思ったが、画像を処理してみると、average poolingは、コントラストが下がり、max poolingはコントラストが上がったので、納得した。
4. The Sliding Window
5. custom Convnets
6. Data Augmentation
Keras lets you augment your data in two ways.
The first way is to include it in the data pipeline with a function like ImageDataGenerator. The second way is to include it in the model definition by using Keras's preprocessing layers. This is the approach that we'll take. The primary advantage for us is that the image transformations will be computed on the GPU instead of the CPU, potentially speeding up training.
これは知らなかった。training timeを短縮できることは重要だ。
model = keras.Sequential([ # Preprocessing preprocessing.RandomFlip('horizontal'), # flip left-to-right preprocessing.RandomContrast(0.5), # contrast change by up to 50% # Base pretrained_base, # Head layers.Flatten(), layers.Dense(6, activation='relu'), layers.Dense(1, activation='sigmoid'), ])
モデルを定義する ところ’model = keras.Sequential’で、最初にimage_transformationの処理、preprocessing.RandomFlip('horizontal')、を行なえばよいということだ。
10月26日(月)
RSNA:
明日の午前9時がsubmissionの締め切りだ。
残念だが、作業は終了としたし、メダル圏内に入る可能性もない。
Courses:
今後は、公開コードのフォークと並行して、自分で作成したコードをもってコンペに参加できるように、Kaggleのコースを最大限、活用していく。
要注意
deep learningのCourseで、notebookを実行するとデフォルトでGPUが立ち上がる場合があり、放置しておくと、使ったことになって、気付かないうちに、10時間くらい消費していた。
GPUを使ってdeep learningのtrainをパラメータを変えながら体験できるようになっているためであり、すぐに使えるコードだということの証明でもある。
Intro to Deep Learning:
知っているつもりで放置していた。あらためて勉強してみよう。
1. A Single Neuron
We could define a linear model accepting three input features ('sugars', 'fiber', and 'protein') and producing a single output ('calories') like so:
model = keras.Sequential([ layers.Dense(units=1, input_shape=[3])
このコースは、tabular dataのみを扱う。
- apply neural nets to two classic ML problems: regression and classification
2. Deep Neural Networks
Be sure to pass all the layers together in a list, like [layer, layer, layer, ...], instead of as separate arguments.
model = keras.Sequential([ # the hidden ReLU layers layers.Dense(units=4, activation='relu', input_shape=[2]), layers.Dense(units=3, activation='relu'), # the linear output layer layers.Dense(units=1), ])
F. Cholletさんのテキストから、ずいぶん変わったなと思う。第2版はまだかいな。
A. Geronさんのテキストは第2版でTF2対応になったし、複数の形式で説明している。
3. Stochastic Gradient Descent
4. Overfitting and Underfitting
full connection modelでも、deepとwideがあるのか。単純に1層あたりのノード数を増やせばwideってあたりまえか。これから意識してみよう。たとえば、線形性が高い場合は幅を拡げる、非線形性が高い場合は深くする。これらは、意識していなかった。
earlystoppingはoverfittingを防ぐものだと思っていたが、underfittingをも防ぐとの記述に目からうろこ。言われてみれば、そうだよな。損失曲線の底付近得を見つけて、その付近で最適モデルになったとみなして、trainingを停止するわけだから。
Early Stopping Callbackは、使い方等がわかったが、GroupShuffleSplitについての説明が見当たらず、全く理解できていない。なぜこの場合に必要なのか、どういう効果があり、どういう作用があるのかなどまったく不明。
5. Dropout and Batch Normalization
10月27日(火)
RSNA:
午前9時00分、Final submission deadline
暫定順位:274位
獲得技術:無
技術向上:無
今後に向けて:おんぼろでもいいから、自前のコードをつくる。
Cources:
Intro to Deep Learning
5. Dropout and Batch Normalization
batch normalizationの意味がよくわかった。
CNNでよく使っていたのだが、それほど大きな効果は認められなかった。
入力データの規格化もできるということは、知らなかった。
6. Binary Classification
layers.Dense(1, activation='sigmoid')
model.compile( optimizer='adam', loss='binary_crossentropy', metrics=['binary_accuracy'], )
Cources:
Feature Engineering
1. Baseline Model
Using this data, how can we use features such as project category, currency, funding goal, and country to predict if a Kickstarter project will succeed?
これらのfeatureからprojectが成功する確率を計算するわけだが、これらのfeatureに、成功の要因がどれだけ含まれているか疑問である。
ラベルは、正解ではなく、結果であり、必然といえる要因が含まれているとは思えない。それでも、ここでは、技術を習得することを優先しないといけない。
Prepare the target column
First we'll convert the state column into a target we can use in a model. Data cleaning isn't the current focus, so we'll simplify this example by:
- Dropping projects that are "live"
- Counting "successful" states as
outcome = 1 - Combining every other state as
outcome = 0
この作業に異論はない。ターゲットは、プロジェクトが成功するかどうかである。
Prep categorical variables
Now for the categorical variables -- category, currency, and country -- we'll need to convert them into integers so our model can use the data. For this we'll use scikit-learn's LabelEncoder. This assigns an integer to each value of the categorical feature.
文字情報を整数に置き換えるのだが、識別子としての意味しかないので、ここに、違和感を感じる。
2. Categorical Encodings
In this tutorial, you'll learn about count encoding, target encoding, and CatBoost encoding.・・・識別子+αの意味を持たせる方法の説明のようだ。
Count encoding replaces each categorical value with the number of times it appears in the dataset. For example, if the value "GB" occured 10 times in the country feature, then each "GB" would be replaced with the number 10.
category, currency, and countryこれらを出現頻度数に置き換えると、AUCが0.7467から0.7486に上がった。
Target encoding replaces a categorical value with the average value of the target for that value of the feature. For example, given the country value "CA", you'd calculate the average outcome for all the rows with country == 'CA', around 0.28.
これは、おかしいな。ターゲットの値を使っている。train_dataだけで、validation_dataとtest_dataに使わなければtarget leakではないと書かれているが、納得できない。
これを使うとAUCは0.7491になるとのこと。
Finally, we'll look at CatBoost encoding. This is similar to target encoding in that it's based on the target probablity for a given value. However with CatBoost, for each row, the target probability is calculated only from the rows before it.
これを使うとAUCは0.7492になるとのことだが、よくわからん。
3. Feature Generation
4. Feature Selection
Lyft:
計算終了後のsaveでHDDが容量オーバーで停止した。なんてこった。
条件を変えて計算中に気がついたら停止していた。今日はついてない。
追加学習と思ってちょっと学習させてみたが、ゼロからの学習だったようで、話にならないことが分かった。なにかやろうとすれば、それだけコードを読みにいくので、まったく無駄な作業ということではない。
約3時間かかって、あと10分から20分くらいで終わるはずのところで、また、妨害された気がする。どうも、サーバー側でなにかやらかしている気がする。
Your notebook tried to allocate more memory than is available. It has restarted.
全く問題ない状態で動いているはずなのに、突然停止した。
腹立つけど、無料だから、文句が言えないな。
午前3時くらいまでかけてcommitする予定だったのだが。
10月28日(水)
Lyft:
Final submission deadline: November 25
今後の予定
1.train済みモデルを3種類作成し、これらのtrain済みモデルのパラメータをkaggle datasetsとして保存した後、このパラメータを読み込んでinferenceする。
とりあえず、不十分だが、trainしたモデルのパラメータをkaggleのdatasetsにして、inferenceしてみる。
trainingにはかなり時間がかかりそうだ。
2.base_modelを変える。
3.画素数を変える。
Cource:
Deep LearningのExerciseは、立ち上げと同時にGPUがOnになるものがある。このとき、別画面でKaggle notebookをGPUで走らせていると、警告なしに、Kaggle notebookは停止する。計算はゼロからやり直しになるので、要注意。
Deep Learning以外のExerciseでも、立ち上げと同時にGPUがOnになるものは、同様の症状が生じるので要注意。
(コンペ間でも、GPU間で衝突すると、計算は即時停止となるので、要注意。)
Feature Engineering
Exercise 1 :
q_1が要求している解答/正解とSolutionが異なっている。
Solutionを使うと、次に進めなくなる。出来上がるDataFrameの名称が異なる。
<雑談>
画像を解釈するとき、ヒトは、全体から細部へ、細部から全体へ、視野を変えながら眺めまわす。こういうことを実現すれば、高精細な画像と大きなモデルの単純な組み合わせに優るものになるかもしれない。自然言語処理においても、ヒトが翻訳したり作文したり抄録を作成したりする過程をモデルに組み込むことができれば・・・。
まだまだ単細胞的な働きしかできていないのかもしれないと思って、モデルの高度化を目指した取り組みをしていかないと、と思うのだが、それを実現するには、プログラミング技術が必要である。プログラミングは、Pythonに限る必要はない。適当なものがなければゼロから言語を創ればいいだけのこと。
画像処理だと、複数の解像度の画像を、複数のモデルで学習/予測するだけでなく、複数の箇所に分割して学習/予測し、複数の標的を探し出し、標的毎に特徴量を抽出し、課題との関連を学習し、・・・。
10月29日(木)
MoA:
さあ、挑もう、と思っても、意味の分からない文字と数字の羅列。
結局、パラメーター最適化にはまってしまう。
10月30日(金)
MoA:
CVの改善を進めるだけなのだが、Lyftのtrainで3日前にGPUを使い果たしたので、tab_dataといえども、feature数が多く、hidden_sizeが比較的大きいNNを使うと、当然であるが、CPUだけでは、時間がかかるが、特定の指標の変化だけなら調べることができて、パラメータの最適化に、ある程度は使える。
NNの1層あたりのユニット層を増やすと、あるところまでは改善され、それ以上では飽和もしくは悪化する。また、NNの層数を増やすと、あるところまでは改善され、それ以上では飽和もしくは劣化する。underfittingとoverfittingの間を動いているだけ。
10月31日(土)
MoA:
GPUが使えるようになったので、お気に入りの条件で計算してsubmitしたら、1400-1500位相当であった。public_dataに無理に合わせるのはダメだと言われても、離れすぎているのもダメだし、Train_data(val_data)に合わせすぎるのもダメだしな。
コンペでは、最終的に、2件の投稿しかできないということの意味が、少しづつわかってきたような気がする。コンペが終わってから、あの結果を選んでおけばよかった、などというのは、だめなんだ。
ついこのあいだも、めんどくさいと思って最新の2件を選んだら、弾き飛ばされて、後でよく見たら、ゆうゆうメダル圏内のものがあった。それをsubmitしたときのメモを探して見返してみたら、NNの条件が偏っていたので、もっと汎用的なモデルにするためにパラメータを変えてみる、というメモ書きがあった。しかしながら88件もsubmitしていたのと、データ整理が悪いのと、そのような計算をしていたことを思い出せなかったことなどによって、適切なモデルを選ぶことができなかった。しかし、こういうのは、後出しじゃんけんでしかないだろう。仮に、そのことを思い出し、メモを見ても、そのときのスコア(public LB)では、60%くらいの順位だから、やはり、選ばなかっただろうと思う。
Lyft:
データ量がハンパないコンペで、私でも使えるコードが公開されると、計算環境の勝負になる。もちろん、トップレベルの人たちの間の競争は、別次元だろうとは思うが。
とりあえず、350kのデータを学習させようとすると、KaggleのGPUで200時間くらいかかりそうだ。貧乏人には手が出せないコンペだな。kaggle kernelを、6時間毎に継いで使おうと思っても、途中データを出力するにはcommitしなければならず、時間が足りない。こういう不満を言うのは、初級レベルだということだ。小さくて速いモデルを探して使うとか、TPUやコラボを使うとか、探せば手段はありそうに思う。さぼっているだけなんだろうな。
GPU:初日に12時間25分も使ってしまった。
明日から11月だ。
6月中旬のPANDAコンペ、TReNDSコンペから、本格的に、Kaggleに参加した。
2019年のAPTOSが最初のKaggleだったと思うが、F. Cholle氏のテキスト片手にモデルを作成し、commit, submissionしたが、エラーばかりでリーダーボードに載らなかった。
その後もいくつか挑戦していたが、モデルを作ることもできず、submissionもできないことが続いた。
コンペに参加してもsubmissionしない、できない状況は、ARCでも同じだった。アルゴリズムを考えることに熱中し、文献も読み、公開コードにも学んでいたが、結局、submissionできずに終わった。
2020年5月8日に、Kaggleのリーダーボードに載ること、を目標に定めた。
最初から公開コードを使って参加することに躊躇もあった。
その躊躇の気持ちは今も変わらないが、公開コードは、非常に高度な内容を含んでおり、これほどのお手本に直に触れることができるというのは、非常に良い経験になると思っている。
はやく自前のコードで勝負できるようになりたいという思いがあるのだが、公開コードでもまだまだずいぶん先にあるように感じるけれども、コンペの後に出てくるトップクラスのコードは、さらに先をいっているので、コンペ後にGitHubやKaggleのコンペサイトに公開されるトップクラスのコードを時間をかけて理解することもやっていかないとだめである。
AIの学習も、多面的にやっていこう。Kaggle以外も含めて、いろんな方法をensembleしながら進もう。
11月につづく