OSIC Pulmonary Fibrosis Progression
Predict lung function decline
今日から、このコンペに挑戦しよう!
目的:予測モデルを1から作れるようになるためのコードの学習。
手段:お手本モデルから、徹底的に学ぶ。
方法:プログラムのすべての行に、初心者にもわかる注釈をつけよう。
目標1:徹底的に注釈をつけたお手本コードを、初学者(自分:me)のために、公開しよう。
目標2:メダル獲得
8月18日
お手本を探そう。
目的は、概要の最後の方に、次のように記述されている。
In this competition, you’ll predict a patient’s severity of decline in lung function based on a CT scan of their lungs.
You’ll determine lung function based on output from a spirometer, which measures the volume of air inhaled and exhaled.
The challenge is to use machine learning techniques to make a prediction with the image, metadata, and baseline FVC as input.
CT画像と、メタデータと、強制肺活量(Forced Vital Capacity)を入力とし、これらの情報を適切に組み合わせて、肺機能の変化を正しく評価できる機械学習プログラムを作ろう。
2つの公開ノートを組み合わせた公開ノートがある。
最初に、フォーク時のまま走らせてsubmitし、その次に、影響の大きそうなパラメータを変更してみたのだが、オリジナル性の高いモデルは、かなり最適化されていて、一見不自然に見える値でも、案外、最適値に近いことが多い。
ただし、難しいのは、グローバル最適値ではなく、局所最適値になっている場合で、このときは、不自然に見えるパラメータを自然な方向に動かしても、評価値が下がることもよくある。
さらに、出力のばらつきには、常に、注意しなければならない。
実際、今日は、適切と思われる公開コードで、いくつかパラメータを変えて最適化しようとしたが、いずれのパラメータについても、最適値は見つけられなかった。
それだけ再現性が良くないということなのか、それとも、すでにグローバル最適化がなされているということなのか。
明日から、コードの解読を進めながら、丁寧に、最適化を行なっていこう。
8月19日
プログラムありきになってしまっている。
もっというと、Kaggleありきになってしまっている。
機械学習、ディープラーニングは、課題解決の手段であって、目的ではない。
もちろん、Kaggleも目的であろうはずはない。
それなのに、メラノーマの検出も、前立腺がんの診断も、目的を十分に理解せぬまま、単に、目の前にあるプログラムを借りてきて、チューニングしてスコア出して、順位争いすることに集中していた。
正確な診断に役立つプログラムを開発するという目的はどこにいったのか。
良いプログラムにめぐりあえたなら、その性能をさらに引き上げるにはどうすれば良いのか、実用化への課題は何なのか、などを考えることが必要だったのではないのか。
スコアを上げるためだけに奔走し、医療の現場で役に立つプログラムを開発するということを、本気になって考え続けることができなかったことを、反省しよう。
OSICのホームページより、以下、引用
“OSIC was created to bring divergent groups together to look at new ways of fighting complex lung disease,” said Elizabeth Estes, the consortium’s executive director. “In addition to utilizing expertise from academia, industry and philanthropy, we wanted to introduce clinicians to the broader artificial intelligence and machine learning community to see if new eyes and new tools could help us move forward, faster. We are excited to see the progress that can be made for patients all over the world.”
OSIC is supported by a myriad of collaborative academic and industry institutions, including founding members Boehringer Ingelheim, Siemens Healthineers, CSL Behring, FLUIDDA, Galapagos, National and Kapodistrian University of Athens, Université de Lyon, Fondazione Policlinico Universitario Agostino Gemelli, and National Jewish Health. All members work in pre-competitive areas for mutual benefit and, most importantly, the benefit of patients.
本当に求められていることは何か。
CT画像からどれだけの情報を引き出せるか。
教師データは正確か。
CT画像は、病気の進行状況を正確に表しているかもしれないが、それに対する値付けは正確ではない可能性がある。FTV(強制肺活量)は正確に測定できるものではない。必ず、ばらつく。実際、train_dataをみると、時間の経過とともに一様に変化しているものはない。ばらつきの大きさを見積もりながら、減衰曲線を評価する必要がある。病気の進行具合は、全体の平均値、よく似た変化曲線の平均の傾向を用いた方が、より正確かもしれない。unsupervised learningとしてCT画像を解析することも必要ではないか。
メラノーマのコンペでは、画像解析が、スコア競争、アンサンブル競争に置き換わってしまっていた。
医療応用では、画像からどれだけの情報を引き出せるかが重要なのだから、スコアではなく、医療診断画像から適切に情報を引き出すための方法(論)を確立することに集中しよう。
8月20日
tf.keras.callbacks.ModelCheckpoint
計算途中で、lossが最小、accが最大、などの条件が成立したときのモデルのパラメータを保存したり、保存したパラメータを呼び出したりする方法を、自分でゼロから作成するプログラムでは、まだ使えていない。
A. Geronさんのテキストや、TensorFlow/KerasのHPなどで勉強しよう。
明日は、書きかけの過去のブログに追記することにより、この部分を学習しよう。
肺のCT像に関して専門家が興味深い議論をしている。CT像に関しては、専門家がこれまでに多くの議論を重ねてきており、対象とする病気の種類によって、どこに着目すべきかは異なるものであり、今回の場合、何に着目したらよいかを考える指針になるようなことを、具体的に紹介している。
ただし、機械学習やディープラーニングで解析する場合に、専門知識をどう生かすかは難しいところである。何をどう学んでどう答えを出してくるか、使う機械学習モデルの性能に期待し、その性能を向上させる方法を模索しているわけで、そこに、専門家の専門領域の解析手法を持ち込む際には、どう持ち込むか、機械学習との役割分担を明確にしながら、慎重に進めていく必要があるように思う。
8月21日
フォークしたモデルのチューニングをしている。
再現性の確認から始めている。
cvとpublic LB、public LBとprivate LBとの乖離のことを考えながら進めることは、実案件でも重要であろうと思われる。単にコンペで失敗した、ということで終わらせないように、モデルの出力を正しく理解し、ばらつきを抑え、信頼性の高い結果を得るための方法を検討しながら、性能を評価する技術を磨いておこう。
Kaggleから、学習コースの案内がきたので、ちょっと覗いてみたら、データの前処理技術だった。Kaggleのコースは非常にわかりやすくかつ具体的な事例に即した内容になっているので、学習効率が高い。過去にいくつか取り組んだコースについても、身につくまで何度でも繰り返し学ぼう。
他のコンペ:birdcall identificationとlandmark recognitionにも参加してみた。my dear watsonにも参加してみようかな。これらに参加することで、視野が広がることを期待している。音声情報の扱い方とか、分類数が極端に多くかつラベル付きデータが非常に少ないものが含まれるとか、自然言語かつ多種類の言語を扱うものである。すぐには理解できなくても、いろいろなデータやモデルを見るのは面白く、経験を積み重ねることで異なる課題の理解が進むこともあるのではないかと思っている。
エポック数の多さに仰天したコードについて、Vall_scoreを落とさず、エポック数をどこまで下げることができるか調べた。最初に同じ条件で計算したときのばらつきを調べた。次に、エポック数を段階的に下げていって、評価値の変化を調べた。その結果、まあまあ常識的な範囲まで下げることができて、評価値は確実に良くなった。ところが、public LBは、明らかに悪くなっていった。ばらつきの範囲を超えていて、この関係は明白であった。以上のことから、エポック数は、public LBを向上させるために最適化していたのではないかと推測される。もちろん、エポック数を下げることがprivate LBを向上させることにつながるかどうかは、全く分からない。しかし、予測性能を上げるということに関しては、他のパラメータとも合わせて調整する必要があるのかもしれない。それらの作業を適切に行うためには、もっと正確にコードを解読する必要がある。
8月22日
Adamの学習率を0.1にしていることに驚いていたら、こんどは、0.00001(1e-5)に出くわした。
1e-5に対して、1e-4と1e-3と1e-6を試してみた。
20エポック程度では、いずれの場合にも殆ど学習できなかった。
3つのカテゴリーに分類するのだが、accが0.33+から殆ど変化しなかった。
この場合の適切なlrは、およそ、1e-5から3e-6の間であった。
これは、My Dear Watsonでのことである。
Kaggle staffらによるTutorial notebookから始めて、スコアの低い公開コードを適当に走らせてみた。
それぞれの公開コードには、それぞれの特徴があり、それぞれの性能があらわれる。
自然言語を扱ったモデルはあまり動かしたことがなかったのだが、最近急速に性能が上がっているようである。
このサイトの公開コードを十分に理解できているわけではないが、扱うデータ量に比例して性能が向上している(スコアが上がる)ことを強く感じた。
8月23日
明日から本格的にコードを組み立てよう。
まずは、できるところまで、自分の計算環境:Windows10, 64bit, Anaconda3のjupyter notebookでコードを組み立てていく。
*いまどきは、Google Colabなんかを使う方が便利そうなので、そうしたいのだが、まだ使い慣れていないので、今は、Anaconda3-jupyter notebookで進める。
このとき、お手本となるコードをjupyter notebookにアップロードしておく。
最初は、EDA、
次に、予測モデル。
8月24日
まずは、metricの学習から。
FVCの標準偏差σ
標準偏差は70以上:σ-clipped=max(σ, 70)
FVCの予測値と真値の差Δ
Δは1000以下:Δ=min(|FVC-true - FVC-predicted|, 1000)
metric = - √2 * Δ/σ-clipped - ln(√2 * σ-clipped)
仮にσ=70, Δ=1000を代入すると、metric ≒ - 24.8
何を評価するか、およそ理解できたところで、公開コードの中からよさそうなEDAを探して、jupyter notebookにアップロードしておいて、それを参考にしながら、自分のnotebookを作っていく。
CT画像の読み込みには、pydicomが必要なので、Anaconda3にインストールする。
conda install -c conda-forge pydicom
コンペデータを読み込んで、Desktopに置こう。
22.35GBあるので、ネット環境にもよるが少し時間がかかる。解凍にも時間がかかる。
すでに何日か前にダウンロードし、解凍してあるので次に進む。
次は、train_dataを読み込もう。
df_train = pd.read_csv('D:/.../.../osic-pulmonary-fibrosis-progression/train.csv')
df_train.shape
(1549, 7)
1549行、7列
7列は、'Patient', 'Weeks', 'FVC', 'Percent', 'Age', 'Sex', 'SmokingStatus'である。
1549行は、1549人のPatientではない。
df_train['Patient'].nunique( )
176
患者の数は176人で、各人について、何週目かにおけるFVC値が、ml単位で、平均して9件程度、掲載されている。
FVCデータ数は、6件が2名、7件が7名、8件が25名、9件が132名、10件が10名である。
teat_dataをみてみよう。
df_test.shape
(5, 7)
なんと、5人分しかない。
その5名の患者の情報の種類は、train_dataとまったく同じだが、FVCは1回分だけしか与えられていない。FVCの経時変化と標準偏差を予測するのであるから当然である。
sample_submission.csvは、どうなっているのだろう。
df_submission.shape
(730, 3)
患者が5名しかいないのに、730行もあるのはなぜだろう。
3列は、それぞれ、Patient_Week, FVC, Confidenceである。
Pationt_Weekは、患者のIDの後に、-12週から+133週までの識別番号が付加されていて、1名の患者に、-12週から133週までの計136週ぶんのFVCの予測値を記入する。
最初に示したmetricsを式にしておこう。
sigma_clipped, delta, Y_true = FVC_true, Y_predicted = FVC_predicted, score = metricを用いて、
sigma_clipped = np.maximum(sigma, 70)
delta = np.minimum(np.abs(y_true - y_predicted), 1000)
score = - np.sqrt(2) * delta / sigma_clipped - np.log(np.sqrt(2) * sigma_clipped)
さて、もっと近づいて、FVCを眺めてみよう。
週単位で測定された 6回から10回のFVCの変化を用いて、12週前から133週後までのFVCを1週間単位で予測することが求められている。
その6回から10回の測定の大半は、60週くらいまでの測定値であるため、求められている最後の3週、131,132、133週の値は、ほぼすべての場合、非常にラフな外挿となるように思う。
test_dataで与えられるのは、tab_dataの他には、どこかの時点でのFVC の値と、CT画像である。
明日は、CT画像について調べよう。
8月25日
train.csv, test.csvの Patient_ID が、DICOMフォーマットで保存されているCT画像のフォルダー名になっている。
train/ には、176のフォルダー、test/ には5つのフォルダーがあり、それぞれのフォルダーの中に、複数の連番のCT画像が含まれている。
train/のデータセットの場合、各フォルダー内の画像の数は、最小で12、最大で1018含まれている。1人の患者に対して1018ものCT画像って、どういうことなのか、さっぱりわからない。
まずは、CT画像を読み込んで、表示してみよう。
CT画像のファイル名は、番号だけで、拡張子が、dcmである。たとえば、1.dcm。
これは、DICOMファイルで、pydicomを使って開くことができる。
pydicomのホームページに、ファイルを読み込んで、画像を表示するための、次のようなコード例が示されている。
>>> import matplotlib.pyplot as plt
>>> import pydicom
>>> from pydicom.data import get_testdata_files
>>> filename = get_testdata_files("CT_small.dcm")[0]
>>> ds = pydicom.dcmread(filename)
>>> plt.imshow(ds.pixel_array, cmap=plt.cm.bone)
わかりやすくするために、dsを、dicom_fileと書き換える。
file_path = 'C:/Users/..../Desktop/osic-pulmonary-fibrosis-progression/train/ID00228637202259965313869/200.dcm'
dicom_file = pydicom.dcmread(file_path)
plt.imshow(dicom_file.pixel_array, cmap=plt.cm.bone)
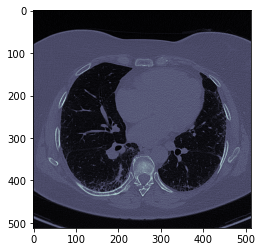
*この画像の解釈:白い部分は骨?:真っ黒い部分は空気なので、黒い部分が肺:肺の中の白い部分は固まった組織?血管?:灰色部分は水分が多く含まれる領域
*HU:空気-1000(黒い):肺-700 to -600:水 0(灰色):血液13 to 50:骨200 to 3000(白い)
このdicom_fileは、このような画像情報に加えて、次のようなメタデータが含まれている。
['BitsAllocated', 'BitsStored', 'BodyPartExamined', 'Columns', 'ConvolutionKernel',
'DeidentificationMethod', 'DistanceSourceToDetector', 'DistanceSourceToPatient',
'FocalSpots', 'FrameOfReferenceUID', 'GantryDetectorTilt', 'GeneratorPower',
'HighBit', 'ImageOrientationPatient', 'ImagePositionPatient', 'ImageType',
'InstanceNumber', 'KVP', 'LargestImagePixelValue', 'Manufacturer',
'ManufacturerModelName', 'Modality', 'PatientID', 'PatientName', 'PatientPosition',
'PatientSex', 'PhotometricInterpretation', 'PixelData', 'PixelRepresentation',
'PixelSpacing', 'PositionReferenceIndicator', 'RescaleIntercept', 'RescaleSlope',
'RescaleType', 'RotationDirection', 'Rows', 'SOPInstanceUID', SamplesPerPixel',
'SeriesInstanceUID', 'SingleCollimationWidth', 'SliceLocation', 'SliceThickness',
'SmallestImagePixelValue', 'SpecificCharacterSet', 'SpiralPitchFactor', 'StudyID',
'StudyInstanceUID', 'TableFeedPerRotation', 'TableHeight', 'TableSpeed',
'TotalCollimationWidth', 'WindowCenter', 'WindowCenterWidthExplanation',
'WindowWidth', 'XRayTubeCurrent']
一人の患者に対して何十枚、何百枚とある画像の中から、どれを選べばよいのだろうか。FVCの変化、病気の進行状態などとの相関性が高い画像をどうやって探せばよいのか。FVCの変化率とCT画像との間に相関があるのだろうか。その相関の有無や大きさをを判定するにはどうすればよいのだろうか。
train/の画像の全体像を知るためには、任意の患者の任意の番号のCT画像を表示できるようにする必要がある。それには、ffmpegが必要だ。
conda install -c conda-forge ffmpeg
その前に、1つのフォルダー内の全ての(一連の) .dcm データを一括して読み込む必要がある。
続きは、明日。
8月26日
今日は、公開コードのEDAで、CT画像の一部を確認した。
症状の違いをCT画像から学習させたいのだが、特定の1枚を抽出するのか、全ての画像を入力してしまうのか、それでCNNに何をどうやって学習させるのかをよく考えないといけない。
FVCとCT画像の相乗効果を出すことが期待されているように思うので、形式的な検討だけではダメ画と思う。
有効な画像を自動で抽出する方法を考える必要があるかもしれない。
フルに使うにしても、画像の枚数が患者によって大きく異なるので、それをどう処理していけばよいのかも考えないといけない。
解析に適したCT画像をどうやって抽出すればよいのかを、CT画像をじっくりながめながら検討しよう。
8月27日
今日は、公開ノートのCT画像を順に眺めてみよう。
tabular_dataの表示の後に、CT画像を表示しているのだが、CT画像とFVCが全く結び付けられずにいる人がたくさんいるように感じた。
症状が現れている画像とそうでない画像を対比して説明しているものがみつからない。
一人の患者に1組のCT画像が与えられていて、それが、正常なのか、症状が現れているのかの判定ができない。
CT画像のどの部分が肺を表しているのかはおよそわかるのだが、どの部分が正常で、どの部分が異常なのかが、まだはっきりとは、わからない。
前進しているのかな。
8月28日
今日は、借り物でチューニングしただけである。
8月29日
借り物を解読しようとしているのだが、進まない。
明日こそ!
8月30日
停滞
8月31日
ここ数日は、Kaggleの他のコンペで遊んでいた。
I'm Something of a Painter Myself - Use GANs to create art - will you be the next Monet? に参加して、公開コードを実行していた。賞金レースではないし、メダルも出ない。教育的なコンペで、チュートリアルコードが用意されている。チュートリアルコードをチューニングしてスコアを上げるのだが、何回かやれば、頭打ちになる。次のコードを探すのだが、つわものがやってきて、ハイスコアのコードを見せてくれる。チュートリアルにはなかった、augmentationを追加しており、見事な出来栄えになっている。ベースコードに機能が追加されているので、両者を比べることによって、何をどこに配置すればよいのかがわかるので、効果を肌で感じながら、コーディング技術を学べる。
チュートリアルコードは、CycleGANを使っている。David Fosterさんの著書にGenerative Deep Learning - Teaching Machines to Paint, Write, Compose and Play - というのがある。第5章のPaintでは、CycleGANが20ページ、Neural Style Transferが10ページほど、紹介されている。自分はまだNeural Style Transferしか使ったことが無いので、これは良い機会だ。
9月1日
進捗なし。
9月2日
1.肺線維症がCT画像上でどのように観察されるのかを示すデータを探す。
2.コンペで提供されているCT画像から、肺線維症であるかどうかを判断するために用いる画像を抽出する方法を検討する。
・全画像を用いるのが良いのか、特定の場所の画像のみ用いるのが良いのか。
・全画像を用いる場合、画像の枚数の極端な違いをそのままにしておいてよいのか。
・特定の画像を用いる場合、適切な画像をどうやって決めるのか。
3.CT画像から、症状の程度を正しく判定するために必要な情報は何か。
・FVCの変化とCT画像との対応はとれるのか。
・FVCの経時変化はtrain_dataとして与えられているが、CT画像は、一時のデータのみ。
・CT画像はいつ測定したものか。
*思いつくまま書いてみたが、train_dataの内容が把握できていないことは明らか。
9月3日
Discussionに学ぶ
CT画像内の肺のセグメンテーションについては、Kaggleの過去のノートブック、GitHubなどを参照すればわかるとのこと。
肺のCT画像から、FVCの減衰率を予測する技術を競うもの。
疫学的要因もFVCの減衰率と相関性は認められるが、補助要因であり、CT画像との相関とは、区別して扱うべきもの。
CT画像とDICOMファイルの情報から、肺のサイズが計算できるので、FVCとの相関がわかる。
FINDING AND FOLLOWING OF HONEYCOMBING REGIONS IN COMPUTED TOMOGRAPHY LUNG IMAGES BY DEEP LEARNING
Emre EGR˘ ˙IBOZ1, Furkan KAYNAR1, Songul VARLI ¨1, Benan MUSELL ¨ ˙IM2, Tuba SELC¸ UK3
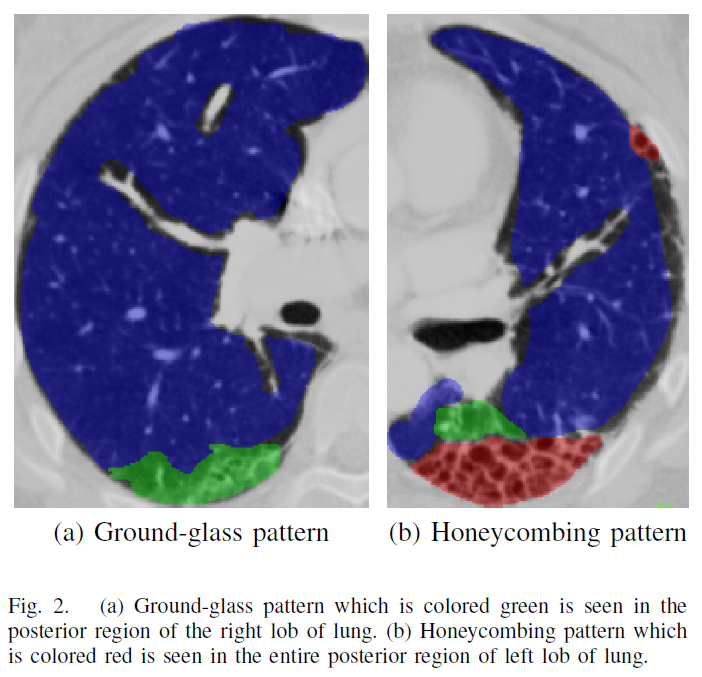
黒い領域は空気もしくは肺胞?:肺の内部に生じている網目状の白い組織が、肺線維症の病巣?

やっと、CT画像中における、肺の構造、線維症の病巣の様子が少しわかったような気がする。
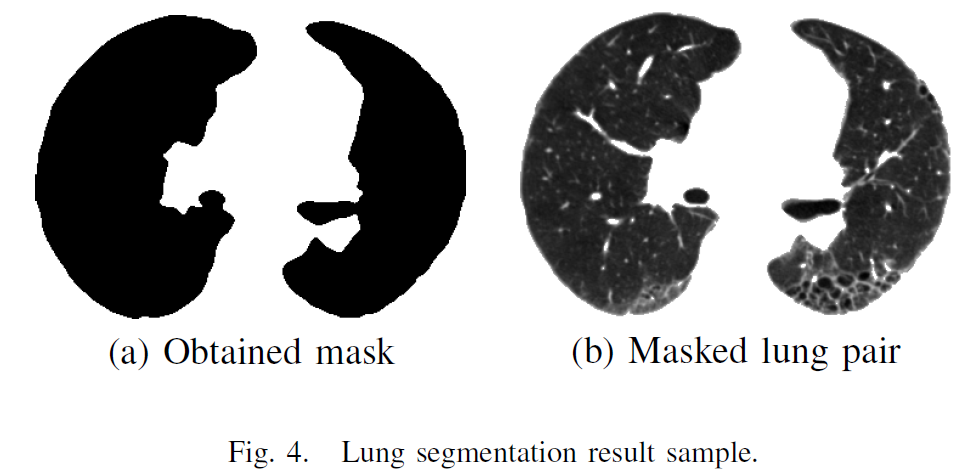
CT画像は、全て用い、その際には、マスクを使うということ。
公開コードは、あらかじめマスクを作成し、Kaggle内のpublicデータとして保存し、呼び出して用いている。最近は、.csvファイル以外は、あらかじめ加工してpublic_dataとして保存して用いるのが普通になっているようである。この方法であれば、外部データもあらかじめ使用許可を得て、public_dataとして保存して、Kaggle-kernelから呼び出せば、internet-offでも問題なく使えるということである。
9月4日(金)
今日は、気になるチューニングのみを行った。
9月5日(土)
今日は、マスクの作り方を学ぼう。
手が回らなかった。
9月6日(日)
進捗なし。
順位が大きく下がっていたので調べたら、また、自分より良いスコアのコードが公開されている。
締切1週間くらい前までは、起こりうること。
目先のスコアを上げようとして、がんばりすぎると、public dataにoverfitしてしまって、蓋を開けたら大きな転落もあるので、難しい。
9月7日(月)
コードの学習が進まないので、次の手を考える。
1.自前のEDAコードを作成する。
2.自前の解析コードを作成する。
ゼロから自前で作る必要はない。お手本を探して、真似すればよい。
手入力の必要もない。コピペでもなんでもよい。
わかりやすいように、編集しなおせば良い。
これで、1ミリでも前進できれば良い。
9月8日(火)
コピペして、変数を確認する、文法を調べる、動作を確認する、などなど、なかなかはかどらない。
進むにしたがって、複雑になり、こんがらがってくる。
ここに書いている時間がない。
9月9日(水)
チューニングで少し前進するが、自分と似たようなスコアの公開コードが現れた。
9月10日(木)
今日もチューニング、つまらない作業だが、つまらなくしている原因は、コードを理解しきれず、自分で本格的なコーディングも本質的な改造もできないことにある。
9月11日(金)
Kaggle Kernel内で、新規にnotebookを用意し、数ある公開notebookの中から、自分に適した筋の良いものを選び出し、新規のnotebookにコピペし、解説を加え、できれば改造もして、機能アップして、再公開する、ということを目的としてここまでやってきたが、殆ど進んでいない。
このやり方の良くないところは、コピペをすること自体にある。コピペの何が悪いのかを考えてみた。
1.コピペ自体が手間である。
2.コードの理解が進まないのは、まとめてコピペするからだ。コードを覚えたければ、タイピングするか、理解できる単位ごとに、区切って、コピペするべきだと思っていたが、今日、ふと、そうではないだろうと思った。
3.コードを覚える瞬間というのは、書式と動作が結びついたときである。書式は見ればわかる。動作は、コードを走らせて、変数がどう変化したのか、書式がどうなっていたのか、書式はどう変化したのか、テンソルの値や書式や次元がどう変化したのか、計算結果はどうなったのかなどを具体的にみることによってしかわからない。
4.動作確認のとき、なぜそうなるかは、与えられたコードをそのまま走らせただけではわからないことが多い。書式や数値や並びや分離記号などを種々変えながら走らせてみることによって、それぞれの意味を理解できるようになる。
5.だとすれば、公開コードはKaggle Kernel上ですぐ動かせるし、編集も自由にできるのだから、新たにnotebookを作る必要などない。
6.新たにnotebookを作るのは、数十分もあれば、自前で、ゼロから、一通りのプログラムを書くことができる人がすることである。
7.新たにnotebookを立ち上げて公開コードをコピペあるいは転載するのは、愚の骨頂である。公開コードを直接編集モードに切り替えて、使えるところは使わせていただいて、不要と思う場所は切り捨てて、追加したいものがあれば、別のコードからコピペすればよいだけである。
8.ただし、以下のことは守らねばならない。
・転載に許可がいる場合は許可を得ること。
・特別に許可が要らない場合でも、引用部分に対しては引用元を明示すること。
・公開する予定が無くても、いつ公開しても良いように、引用元は明示し、変更部分はその概要を明示すること。
・編集モードにしたときは、必ず、引用元を最上部に記入しよう。
9月12日(土)
チューニングは、軽んじてはいけないと思うこともある。
コンペでは、Public dataに対してチューニングするしかないのだが、そうすると、Public dataに対して、overfitthingに向かうはずである。
Public dataに完璧にチューニングしたモデルが、Private dataに対しても完璧にチューニングされるということはありえない。
ではどうするか。
9月13日(日)
チューニングの仕方を深く学ぶことは重要なんだろうなと思う。
9月14日(月)
Kaggleの他のコンペだけど、公開コードを借りた人が、重要な改善点を見つけてスコアを上げ、それを公開した。
そうすると、元の作成者は、改善点を見つけた人に礼を述べるとともに、その改善を行い、その改善内容について説明を加え、再度公開していた。
このように、公開コードを読み込んで、作成者の気付かないところを改善したり、改善や変更の提案などのやりとりに加われるようになりたいものだ。
(この世界では、提案ばかりして、やってみせないでいると、きらわれるようだ。実効性は課題によって違うので、実施した結果とともに示すことが求められている。)
9月15日(火)
今使っているコードは、不自然なことが多い。
その原因は、Lossが小さく、評価も良い結果が、LBの向上につながらず、LBスコアが下がってしまう。
LBスコアを上げるために、パラメータを、不自然な方向に変更しているように感じている。
9月16日(水)
public dataにオーバーフィットさせて、失敗を繰り返しているので、public dataのLBスコアに振り回されることのないように、もっと、よく考えながら、進めよう。
9月17日(木)
スコアの高い公開コードで計算してみた。
GPUの残り時間が少なかったので、GPU使用時間削減を最優先とした。
そのため、前半の9回の繰り返し計算を1回とし、後半は何も変更せず計算した結果、PublicLBスコアは、オリジナルでの値よりは低いが、その違いは予想外に小さかった。
その理由は、前半と後半の重みが後半に偏っているためである。
それにしても、漫然と9回も繰り返し計算していたのは何だったのかと思う。
9月18日(金)
今日は、Landmarkコンペにかかりっきり。
9月19日(土)
この記事はここで終了する。
*反省
1.手本になる公開コードが見つからず(コードを理解しきれない)注釈付与ができなかった。
2.CT画像から、症状の程度やFVCの減少を推定することができるようなモデルをつくろうと考えていたが、できなかった。
3.使っている公開コードのチューニングはある程度までは進んだが、モデルの理解が不十分であり、モデルを改善する方向が途中で見えなくなった。
4.個別モデルのval_lossの減少方向とpublic LBが逆相関になり、解消方法がわからない。CT画像の重みに対する疑問が残ったままである。
このコンペが終わったら、追記する予定。
以上


