Predicting Molecular Properties-2
*1週間程度の予定で、過去のコンペ、Predicting Molecular Properties、に取り組む。
*目的は、DNNがどのような課題に対して、どのように貢献できるのかを、実例を通して学ぶこと。
*今日は、分子構造から分子の量子力学的性質を予測するような文献の調査を行う。
・昨日の2件の文献は、コンペのテーマと同じ内容であったが、文献数が少ないので、検索範囲を拡げる。
*関連文献
Building attention and edge message passing neural networks for bioactivity and physical-chemical property prediction
M. Withnall, E. Lindelof, O. Engkvist and H. Chen
J Cheminform (2020) 12:1
・QSAR:Quantitative Structure Activity Relationships
・Researchers have proposed numerous descrtors to represent molecular 2D and 3D structures, aiming to correlate these descriptors with predicted endpoints.
・Approaches to generating representations using the graph representation of a molecule include graph kernels, and perhaps most importantly in the present context, ECFP (Extended Connectivity Circular Fingerprints).
・Recentry, deep neural network methods have become the latest weapon in a Cheminformatician's arsenal for doing QSAR.
・This rapid growth is due in part to the substantial increase in available biochemical data thanks to the rise of techniques such as High Throughput Screening (HTS) and parallel synthesis, and also to the recent surge in parallel computational power that can be feasibly attained by harnessing General Purpose computing on Graphics Processing Units (GPGPU).
・Efforts have also been taken to enable neural networks to do representation learning, i.e. the neural network is able to learn descriptors itself instead of relying on predefined molecular descriptors.
・Among these, the graph convolution network (GCN) is gaining popularity and various architectures have been proposed in data science community.
・Graph Convolutional Network (GCN), Graph Attention Network, Graph Autoencoders, Graph Spatial-Temporal Networks, ・・・
・In order to cope with the irregurality of graph data, alternative approaches must be designed, most notably to circumvent the issue of irregular non-Euclidian data, and to be invariant to the graph representation.
・Recently researchers from Google put forward an new NN architecture called as message passing neural networks (MPNNs) and used the MPNNs to predict quantum chemical properties.
・The MPNN framework containes three common steps:
(1) message passing step, where, for each atom, features (atom or bond features) from its neighbours are propagated, based on the graph structure, into a so called a message vector;
(2) update step, where embedded atom features are updated by the message vector;
(3) aggregation step, where the atomic features in the molecule are aggregated into the molecule feature vector.
・These molecule feature vector can then be used in a dense layer to correlate with the endpoint property.
・分子の情報をコンピュータに認識させるために、どう記述するかということは、機械学習を適用して、分子の物理化学的、量子化学的、分子生物学的情報を得るために、非常に重要であり、多くの研究がなされてきたようである。
・第一原理計算によって、分子中の各原子の座標が決まれば、分子の物理化学的性質が高い精度で計算できるようになってきたが、時間とコストがかかるので、第一原理計算の役割をニューラルネットに置き換えることが進んできた。
・分子の情報は、物質によっては、原子の座標だけでは表せないので、表現方法に対応したニューラルネットが開発されてきた。
・MPNN(message passing neural networks)はそのうちの1つである。
*関連文献
MoleculeNet: a benchmark for molecular machine learning
Z. Wu, B. Ramsundar, E. N. Feinberg, J. Gomes, C. Geniesse, A. S. Pappu, K. Leswing and V. Pande,
Chem. Sci., 2018, 9, 513

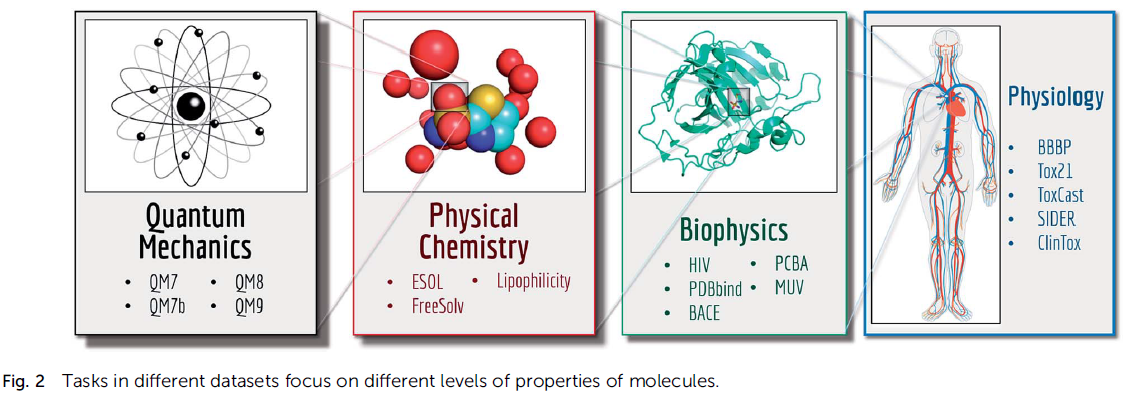

*MoleculeNetの機能を表す3枚の図を示した。
*以下に、キーワードを列挙する。
・Datasets: QM7/QM7b, QM8, QM9, ESOL, FreeSolv, Lipophilicity, PCBA, MUV, HIV, PDBbind, BACE, BBBP, Yox21, ToxCast, SIDER, ClinTox,
・Featurization: ECFP, Coulomb matrix, Grid featurizer, Symmetry function, Graph convolutions, Weave,
・Models - conventional models: Logistic regression, Support vector classification, Kernel ridge regression, Random forests, Gradient boosting, Multitask/singletask network, Bypass multitask networks, Influence relevance voting,
・Models - graph based models: Graph convolutional models (GC), Weave models, Directed asyclic graph models (DAG), Deep tensor neural networks (DTNN), ANI-1, Message passing neural networks (MPNN)
*関連文献
Neural Message Passing for Quantum Chemistry
J. Gilmer, S. S. Schenholz, P. F. Riley, O. Vinyals and G. E. Dahl
2017, arXiv preprint arXiv:1704.01212.

・MPNNs (Message Passing Neural Networks)の仲間とみなされる6種類のNNを束ねて一般化したモデルを提案するもので、各モデルで用いられている式を比較しながら議論が進められている。
・ちゃんと理解したいものだ。
<追記>
*この論文およびMPNNを今回は本命モデルとする。
*関連文献(論文タイトルの最後のgraph networksを見て、関連文献と判断)
Relational inductive biases, deep learning, and graph networks
P. W. Battaglia et al., arXiv:1806.01261v3 [cs.LG] 17 Oct 2018
<追記>
*この論文は、2018年の時点での、Graph Networkに関する非常に優れた解説ではないかと思う。
*以上で、文献調査を終了する。
・MoleculeNetは、GitHubにDeepChemというコードが掲載されているようなので、どこかで集中的に取り組んでみたいと思う。
・このコンペのコードとしては、Neural Message Passing for Quantum Chemistryが本命かなと思うので、MPNNを使っているnotebookがあれば、それをお手本として、明日から勉強しよう。
*明日は、MPNNを使ったコードをお手本にしてプログラミング技術を学ぶ。
つづく


